상세 컨텐츠
본문
작성자: 13기 김현지
타이타닉 데이터 셋을 활용해 Binary Classification 문제를 해결하기 위한 데이터 분석 과정을 공부해 본다.
- 데이터 셋: Titanic - Machine Learning from Disaster
- 참고 커널:
- 커널 필사: KUBIG 코드 분석 스터디 Github → 2주차 → 타이타닉 코드들
타이타닉 데이터를 활용한 이진분류(커널 필사 내용 요약, 정리)
캐글 커널 내용을 요약, 정리한 글입니다. 위 Github 코드를 참고하여 같이 봐주세요:)
Binary Classification Process
- 데이터 셋 확인
- 탐색적 데이터 분석
- 피쳐 엔지니어링
- 모델 만들기
- 모델 학습 및 예측
- 모델 평가
1. 데이터 셋 확인
타이타닉 데이터 셋
| 변수 | 정의 | 설명 | 타입 |
| survived | 생존여부 | target label. 1, 0으로 표현됨 | integer |
| Pclass | 티켓의 클래스 | 1=1st, 2=2nd, 3=3rd로 나뉨, categorical | integer |
| sex | 성별 | male, female로 구분, binary | string |
| Age | 나이 | continuous | integer |
| sibSp | 함께 탑승한 형제와 배우자의 수 | quantitative | integer |
| parch | 함께 탑승한 부모, 아이의 수 | quantitative | integer |
| ticket | 티켓 번호 | alphabat + integer | string |
| fare | 탑승료 | continuous | float |
| cabin | 객실 번호 | alphabat + integer | string |
| embarked | 탑승 항구 | C = Cherbourg, Q = Queenstown, S = Southampton |
string |
- Feature: Pclass, Age, SibSp, Parch, Fare 등
- Target label: Survived
describe() 메서드를 활용한 데이터 셋 확인
다양한 통계량을 요약해 주는 메서드
df_train.describe()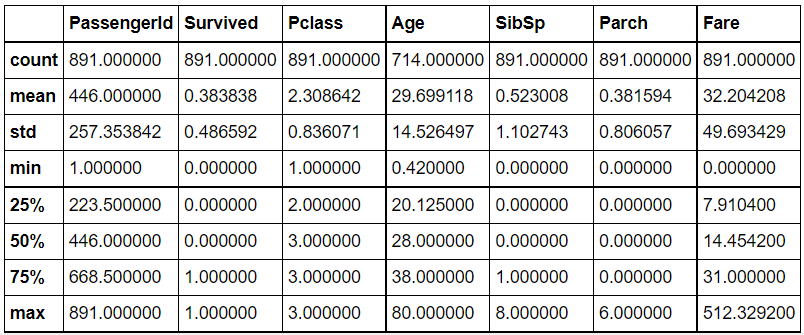
- series에 대한 요약이 수행된다.
- Dataframe의 경우 열에 대한 요약이 수행된다.
- 기본적으로 누락데이터(NaN)는 제외하고 데이터 요약이 수행된다.
Null data check
isnull() 메서드를 활용한 결측치 확인
df_train.isnull()결측값은 True로, 결측값이 아닌 값을 False로 반환한다.
df_train.isnull().sum()각 컬럼마다 결측값이 몇 개인지 확인하기 위해 sum()함수를 덧붙여 사용한다.
Missingno 라이브러리를 활용한 결측치 시각화
결측치를 시각화할 수 있는 모듈이 있는 라이브러리
기본 내장이 아니기 때문에 직접 설치해야 한다.
# 라이브러리 설치
!pip install missingno
# 모듈 임포트
import missingnp as msno- matrix
msno.matrix(df=df_train.iloc[:,:], figsize=(8, 8), color=(0.8, 0.5, 0.2))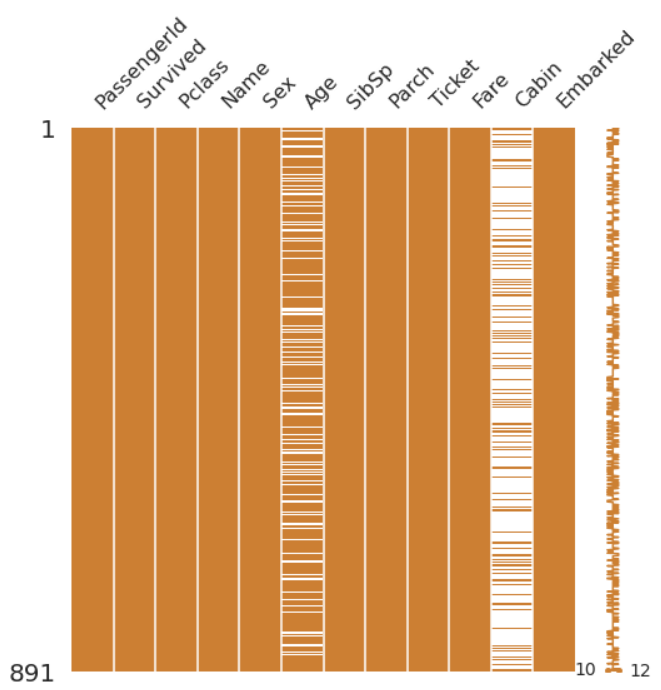
- Bar Chart
msno.bar(df=df_train.iloc[:, :], figsize=(8, 8), color=(0.8, 0.5, 0.2))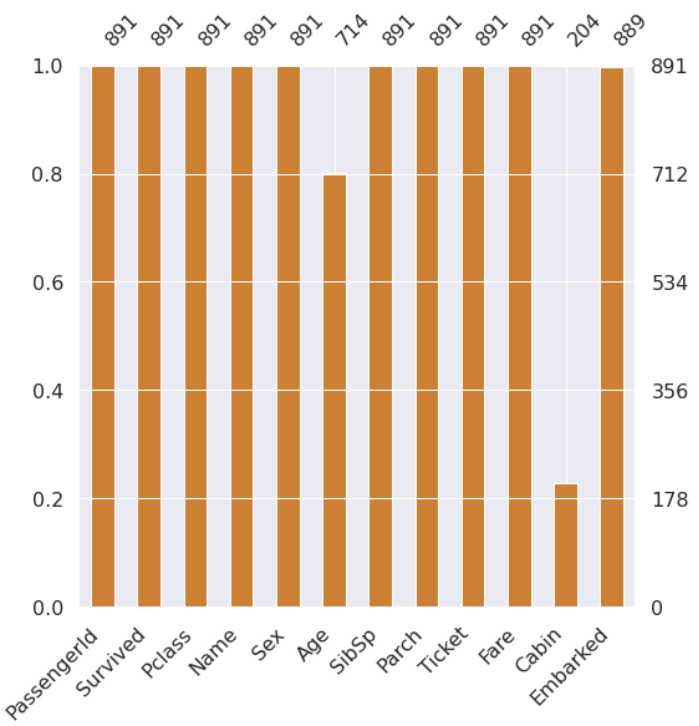
Target label 확인
예측하고자 하는 Target label의 분포가 균일한지, 불균형한 데이터 셋인지 확인한다.
value_counts() 메서드를 활용한 분포 확인
요소별 개수를 확인할 수 있는 메서드
df_train['Survived'].value_counts()
- 시각화를 통해 분포를 확인한다.
f, ax = plt.subplots(1, 2, figsize=(18, 8))
df_train['Survived'].value_counts().plot.pie(explode=[0, 0.1], autopct='%1.1f%%', ax=ax[0], shadow=True)
ax[0].set_title('Pie plot - Survived')
ax[0].set_ylabel('')
sns.countplot('Survived', data=df_train, ax=ax[1])
ax[1].set_title('Count plot - Survived')
plt.show()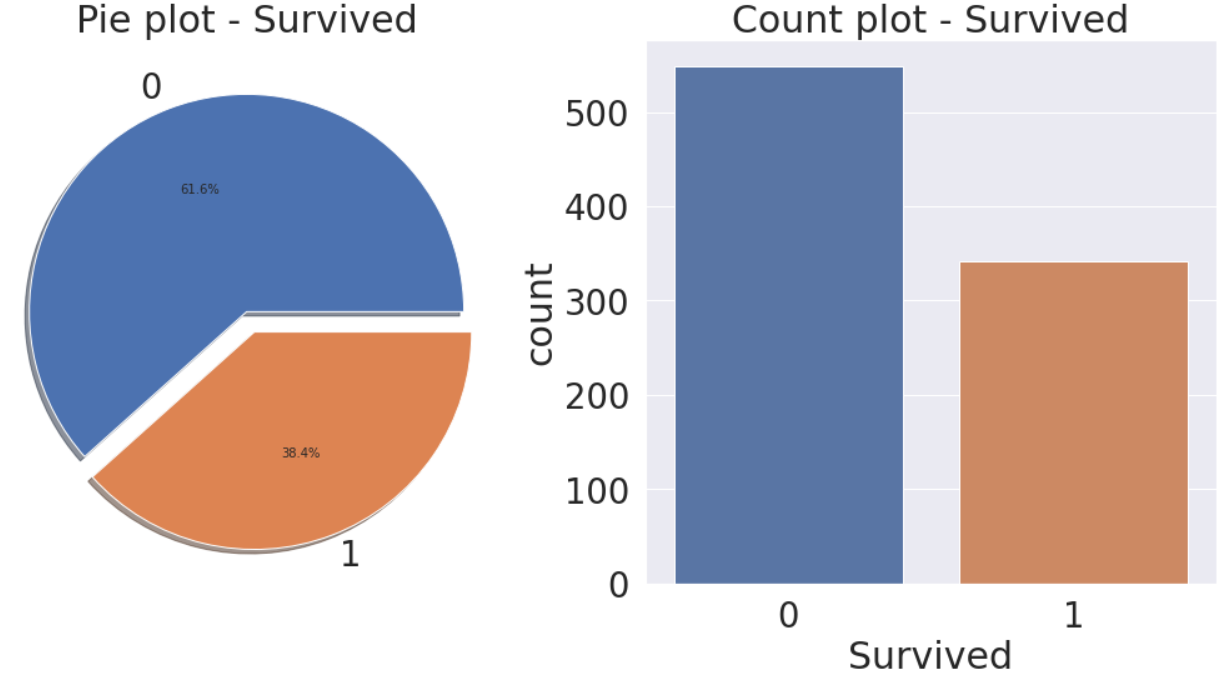
이 데이터 셋에서 target label의 분포는 제법 균일(balance)하지만 불균형한 경우 모델링을 할 때 많은 신경을 써야 한다.
2. Exploratory data analysis
많은 데이터 안에 숨겨진 사실을 찾기 위해서는 적절한 시각화가 필요한데, 시각화 라이브러리 중 특정 목적에 맞는 소스코드를 정리해 두어 필요할 때마다 참고하면 편하다.
- 시각화 라이브러리: matplotlib, seaborn, plotly 등
데이터는 크게 범주형/수치형으로 나뉘고, 이후 각각 명목, 순서, 불연속, 연속 데이터로 세분화 된다.
Oridinal(순서형) 데이터
순서가 있는 범주형 데이터 - Pclass
순서형 데이터의 경우 범주별 데이터의 분포를 확인해봐야 한다.
groupby() 메서드를 활용한 범주별 분포 확인
df_train[['Pclass', 'Survived']].groupby(['Pclass'], as_index=True).count()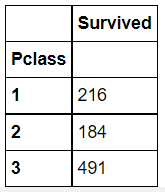
- 위 코드는 'Pclass', 'Survived'로 그룹을 나누고 'Pclass' 데이터에 대한 빈도수 값을 확인하는 코드이다.
pd.crosstab을 활용한 빈도표 생성
데이터프레임의 칼럼(Series 데이터타입)를 파라미터로 넘겨주면 빈도표를 만들어준다.
pd.crosstab(df_train['Pclass'], df_train['Survived'], margins=True).style.background_gradient(cmap='summer_r')
- 시각화를 통해 범주별 데이터 분포를 확인한다.
y_position = 1.02
f, ax = plt.subplots(1, 2, figsize=(18, 8))
df_train['Pclass'].value_counts().plot.bar(color=['#CD7F32','#FFDF00','#D3D3D3'], ax=ax[0])
ax[0].set_title('Number of Passengers By Pclass', y=y_position)
ax[0].set_ylabel('Count')
sns.countplot('Pclass', hue='Survived', data=df_train, ax=ax[1])
ax[1].set_title('Pclass: Survived vs Dead', y=y_position)
plt.show()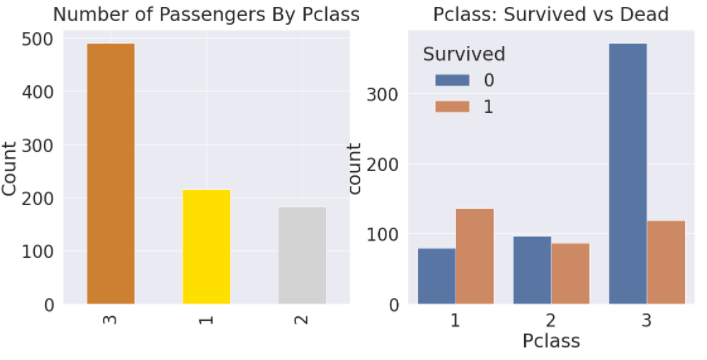
순서형 데이터 EDA: 범주별 데이터 분포 확인을 통해 target label에 대해 해당 변수가 큰 영향을 미치는지 판단해보고, 나중에 모델을 세울 때 해당 변수를 사용하면 좋을지 고민해본다.
Nomial(명목형) 데이터
순서가 없는 범주형 데이터 - Sex
명목형 데이터 EDA: 순서형 데이터와 마찬가지로 범주별 데이터 분포 확인을 통해 target label에 대해 해당 변수가 큰 영향을 미치는지 판단해보고, 나중에 모델을 세울 때 해당 변수를 사용하면 좋을지 고민해본다.
범주형 데이터 2개
factorplot을 활용한 다차원 분석
sns.factorplot('Pclass', 'Survived', hue='Sex', data=df_train, size=6, aspect=1.5)
- Pclass에 대한 생존율을 Sex에 따라 볼 수 있다.
sns.factorplot(x='Sex', y='Survived', col='Pclass', data=df_train, satureation=.5, size=9, aspect=1)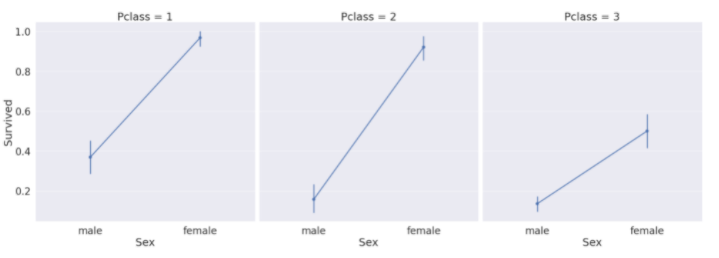
- Pclass 안에서 성별에 따른 생존율을 비교해 볼 수 있다.
Continous(연속형) 데이터
연속되는 수치형 데이터 - Age, Fare
히스토그램을 그려 해당 변수에 따른 밀도를 파악해본다.
sns.kdeplot을 활용한 히스토그램 그리기
fig, ax = plt.subplots(1, 1, figsize=(9, 5))
sns.kdeplot(df_train[df_train['Survived'] == 1]['Age'], ax=ax)
sns.kdeplot(df_train[df_train['Survived'] == 0]['Age'], ax=ax)
plt.legend(['Survived == 1', 'Survived == 0'])
plt.show()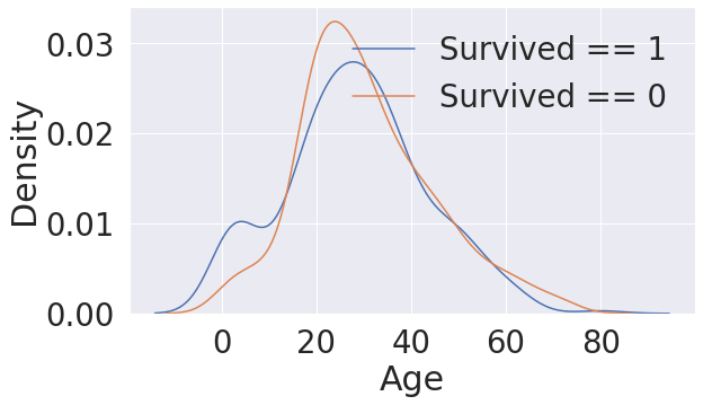
분포가 비대칭한 경우 log 변환해주기

- 특정 피쳐에 대한 분포가 매우 비대칭일 경우, 이대로 모델에 넣어준다면 모델이 잘못 학습할 수도 있다.
- 몇 개 없는 이상치에 대해서 너무 민감하게 반응한다면, 실제 예측할때 좋지 못한 결과를 가져올 수 있기 때문이다.
map 메서드와 lambda 함수를 이용해 컬럼 전체에 로그 취하기
series.map(func) -> data['col1'].map(func)
map함수는 파이썬의 내장 함수로 여러 개의 데이터를 한 번에 다른 형태로 변환하기 위해 사용된다.
- 시리즈에만 적용가능하다.
- 데이터프레임 한 열에 같은 함수를 적용할 수 있다.
lambda 매개변수들 : 식1 if 조건식 else 식2
- 예) lambda x: str(x) if x % 3 == 0 else x → 요소가 3의 배수일 때는 str(x)로 요소를 문자열로 만들어서 반환하고, 2의 배수가 아닐 때는 x로 요소를 그대로 반환한다.
df_train['Fare'] = df_train['Fare'].map(lambda i: np.log(i) if i > 0 else 0)
df_test['Fare'] = df_test['Fare'].map(lambda i: np.log(i) if i > 0 else 0)- 'Fare'열의 값이 0보다 크면 np.log()를 취한다.
연속형 데이터 EDA: 히스토그램을 그려 해당 변수에 대한 분포를 확인해보고, 수치가 변함에 따라 target label이 어떻게 변하는지 파악해본다. 데이터의 분포가 비대칭일 경우 적절한 조치를 취해준다.
범주형 데이터 2개 + 연속형 데이터 1개
sns.violinplot을 활용한 다차원 분석
f,ax=plt.subplots(1,2,figsize=(18,8))
sns.violinplot("Pclass","Age", hue="Survived", data=df_train, scale='count', split=True,ax=ax[0])
ax[0].set_title('Pclass and Age vs Survived')
ax[0].set_yticks(range(0,110,10))
sns.violinplot("Sex","Age", hue="Survived", data=df_train, scale='count', split=True,ax=ax[1])
ax[1].set_title('Sex and Age vs Survived')
ax[1].set_yticks(range(0,110,10))
plt.show()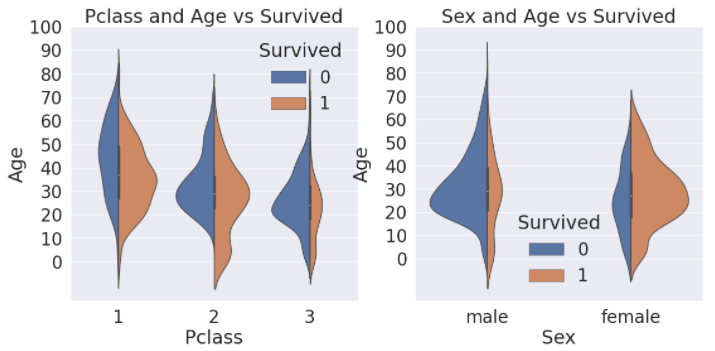
- x축은 나눠서 보고싶은 case(여기서는 Pclass, Sex)로 두고, y축은 보고싶은 distribution(Age)으로 둔다.
- 왼쪽 그림은 Pclass 별로 Age의 distribution이 어떻게 다른지, 거기에 생존여부에 따라 구분한 그래프이고, 오른쪽 그림은 Sex별로 생존에 다른 Age의 분포가 어떻게 다른지 보여주는 그래프이다.
3. Feature engineering
Feature engineering은 실제 모델의 학습에 쓰려고 하는 것이므로, train 뿐만 아니라 test에도 똑같이 적용해주어야 한다!
Fill Null
결측치를 채우는 방법은 정말 많이 존재한다. statistic를 활용하는 방법도 있고, 결측치가 없는 데이터를 기반으로 새로운 머신러닝 알고리즘을 만들어 예측해서 채워넣는 방식도 있다.
여기서 statistics는 train data의 것을 의미한다. 우리는 언제나 test를 unseen으로 둔 상태로 나둬야 하며, train에서 얻은 statistic를 기반으로 test의 결측치를 채워줘야한다.
loc + boolean + column을 사용해 값 치환하기
df.loc["row","column"]
- 첫번째 인자에는 인덱싱할 row를 넣고, 두 번째에는 인덱싱할 column을 넣으면 된다.
df_train.loc[(df_train.Age.isnull())&(df_train.Initial=='Mr'),'Age'] = 33
fillna() 함수로 결측치 채우기
df.fillna(단일값)
- df.fillna(0): 결측치를 0으로 채운다.
df.fillna(method='pad'), df.fillna(method='bfill')
- 'pad': 각각의 결측치 바로 앞에 있는 값으로 채워 넣는다.
- 'bfill': 결측치의 바로 뒤의 값으로 채워넣는다.
Continuous to Categorical
연속형 데이터를 그대로 모델에 쓸 수 있지만, 몇 개의 group으로 나누어 범주화 시켜줄 수도 있다. 연속형 데이터를 범주형 데이터로 바꾸면 정보의 손실이 생길 수도 있으므로 적절하게 사용해야 한다.
- Age 변수 10살 간격으로 나누기
df_train['Age_cat'] = 0
df_train.loc[df_train['Age'] < 10, 'Age_cat'] = 0
df_train.loc[(10 <= df_train['Age']) & (df_train['Age'] < 20), 'Age_cat'] = 1
df_train.loc[(20 <= df_train['Age']) & (df_train['Age'] < 30), 'Age_cat'] = 2
df_train.loc[(30 <= df_train['Age']) & (df_train['Age'] < 40), 'Age_cat'] = 3
df_train.loc[(40 <= df_train['Age']) & (df_train['Age'] < 50), 'Age_cat'] = 4
df_train.loc[(50 <= df_train['Age']) & (df_train['Age'] < 60), 'Age_cat'] = 5
df_train.loc[(60 <= df_train['Age']) & (df_train['Age'] < 70), 'Age_cat'] = 6
df_train.loc[70 <= df_train['Age'], 'Age_cat'] = 7
apply 메서드로 범주화 하기
df.apply(func), df['col1'].apply(func)
- apply 메서드를 사용해 한 컬럼에 함수를 한번에 적용한다.
def category_age(x):
if x < 10:
return 0
elif x < 20:
return 1
elif x < 30:
return 2
elif x < 40:
return 3
elif x < 50:
return 4
elif x < 60:
return 5
elif x < 70:
return 6
else:
return 7
df_train['Age_cat_2'] = df_train['Age'].apply(category_age)- Age의 값이 10보다 작으면 0값, 10이상 20 미만이면 1값 ... 을 반환한다.
String to Numerical
map 메서드를 활용해 사전 순서대로 정리하여 매핑
df_train['Initial'] = df_train['Initial'].map({'Master': 0, 'Miss': 1, 'Mr': 2, 'Mrs': 3, 'Other': 4})- master은 0, miss는 1, mr는 2 등으로 매핑한다.
상관관계 확인
sns.heatmap으로 피쳐들간의 상관관계 분석하기
heatmap_data = df_train[['Survived', 'Pclass', 'Sex', 'Fare', 'Embarked', 'FamilySize', 'Initial', 'Age_cat']]
colormap = plt.cm.RdBu
plt.figure(figsize=(14, 12))
plt.title('Pearson Correlation of Features', y=1.05, size=15)
sns.heatmap(heatmap_data.astype(float).corr(), linewidths=0.1, vmax=1.0,
square=True, cmap=colormap, linecolor='white', annot=True, annot_kws={"size": 16})
del heatmap_data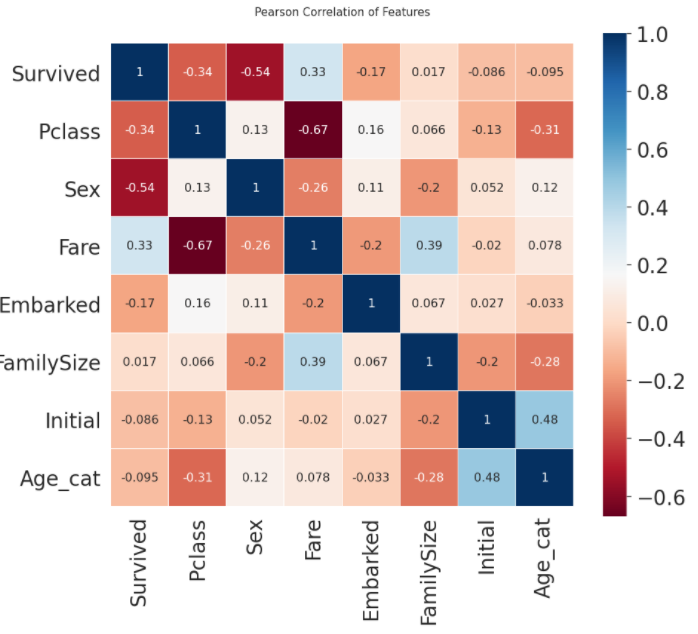
- 1 또는 -1 상관관계를 가지는 feature A, B가 있다면 우리가 얻을 수 있는 정보는 사실 하나일 것이다. 따라서 불필요한 feature가 있을 수도 있는데, 이 경우 모델을 가볍게 하기 위해 피쳐를 제거할 필요가 있다.
One-hot encoding
수치화 시킨 카테고리 데이터를 그대로 모델에 사용해도 되지만, 모델의 성능을 높이기 위해 원핫인코딩을 해줄 수 있다.
- one-hot encoding: 카테고리를 아래와 같이 (0, 1)로 이루어진 n차원 벡터로 나타내는 것이다.
| Initial_Master | Initial_Miss | Initial_Mr | Initial_Mrs | Initial_Other | |
| Master | 1 | 0 | 0 | 0 | 0 |
| Miss | 0 | 1 | 0 | 0 | 0 |
| Mr | 0 | 0 | 1 | 0 | 0 |
| Mrs | 0 | 0 | 0 | 1 | 0 |
| Other | 0 | 0 | 0 | 0 | 1 |
pd.get_dummies를 이용해 one-hot encoding해주기
pd.get_dummies(데이터프레임, columns=[칼럼명1. 칼럼명2, ...], prefix= )
- 첫 번째 인자에는 가변수로 바꾸어줄 데이터를 넣어주고, columns에는 가변수로 바꿔줄 범주형 칼럼 이름을 넣어준다.
- prefix: 생성할 dummy variable의 column 이름 앞에 붙을 prefix를 지정한다.
df_train = pd.get_dummies(df_train, columns=['Initial'], prefix='Initial')
가끔 범주하 100개가 넘어가는 경우가 있는데, 이때 one-hot encoding을 사용하면 column이 100개가 생겨, 학습시 매우 버거울 수 있다. 이런 경우는 다른 방법을 사용해야 한다.
Drop columns
모델에 사용할 column만 남기고 다 지운다.
4. Building machine learning model and prediction using the trained model
EDA를 통해 몇 가지 인사이트를 얻었지만, 이것만으로는 승객이 생존할 것인지 사망할 것인지 정확하게 예측할 수 없다. 이제 몇 가지 훌륭한 분류 알고리즘을 사용해서 승객의 생존 여부를 예측해보자.
지금 타이타닉 문제는 target class(survived)가 있으며, target class는 0, 1로 이루어져 있는 binary classification 문제이다.
Predictive Modeling
train_test_split 모듈을 활용한 데이터 셋 분리
train, test = train_test_split(data, test_size=0.3, random_state=0, stratify=data['Survived'])- test_size: test set(validation set) 구성의 비율
- feature인 X와 target label인 y로 분리한다.
train_X = train[train.columns[1:]]
train_Y = train[train.columns[:1]]
test_X=test[test.columns[1:]]
test_Y=test[test.columns[:1]]
X=data[data.columns[1:]]
Y=data['Survived']
Scikit-Learn 패키지 사용하기
서브 패키지
Scikit-learn은 여러 서브 패키지 단위로 별도의 기능을 제공한다. 이번에 사용할 머신러닝 모형 및 모형 평가 패키지 중 대표적인 패키지는 다음과 같다.
- 모형
- sklearn.svm: support vector machines
- sklearn.naive_bayes: naive bayes
- sklearn.ensemble: Ensemble Methods
- 모형 평가
- sklearn.metrics: Metrics
- sklearn.cross_validation: Cross Validation
- sklearn.grid_search: Grid Search
클래스
원하는 클래스 객체를 생성해서 사용할 수 있다. 대표적인 클래스 그룹은 다음과 같다.
- 머신러닝 모형 클래스 그룹
- Regressor 클래스: 회귀분석
- Classifier 클래스: 분류
- Cluster 클래스: 클러스터링
- 공통 메서드:
- fit(): 모형 계수 추정, 트레이닝
- predict(): 주어진 x값에 대해 y 예측
- score: 성과분석
Radial Support Vector Machine(rbf-SVM)
model = svm.SVC(kernel='rbf', C=1, gamma=0.1)
model.fit(train_X, train_Y)
prediction1 = model.predict(test_X)
print('Accuracy for rbf SVM is ',metrics.accuracy_score(prediction1,test_Y))>> Accuracy for rbf SVM is 0.835820895522388
Linear Support Vector Machine(linear-SVM)
model = svm.SVC(kernel='linear', C=0.1, gamma=0.1)
model.fit(train_X, train_Y)
prediction2 = model.predict(test_X)
print('Accuracy for linear SVM is',metrics.accuracy_score(prediction2,test_Y))>> Accuracy for linear SVM is 0.8171641791044776
Logistic Regression
model = LogisticRegression()
model.fit(train_X,train_Y)
prediction3=model.predict(test_X)
print('The accuracy of the Logistic Regression is',metrics.accuracy_score(prediction3,test_Y))>> The accuracy of the Logistic Regression is 0.8134328358208955
Decision Tree
model=DecisionTreeClassifier()
model.fit(train_X,train_Y)
prediction4=model.predict(test_X)
print('The accuracy of the Decision Tree is',metrics.accuracy_score(prediction4,test_Y))>> The accuracy of the Decision Tree is 0.8134328358208955
K-Nearest Neighbors(KNN)
model=KNeighborsClassifier()
model.fit(train_X,train_Y)
prediction5=model.predict(test_X)
print('The accuracy of the KNN is',metrics.accuracy_score(prediction5,test_Y))>> The accuracy of the KNN is 0.832089552238806
- KNN의 n값을 변화시켜보자.
a_index=list(range(1,11))
a=pd.Series()
x=[0,1,2,3,4,5,6,7,8,9,10]
for i in list(range(1,11)):
model=KNeighborsClassifier(n_neighbors=i)
model.fit(train_X,train_Y)
prediction=model.predict(test_X)
a=a.append(pd.Series(metrics.accuracy_score(prediction,test_Y)))
plt.plot(a_index, a)
plt.xticks(x)
fig=plt.gcf()
fig.set_size_inches(12,6)
plt.show()
print('Accuracies for different values of n are:',a.values,'with the max value as ',a.values.max())
>> Accuracies for different values of n are: [0.75746269 0.79104478 0.80970149 0.80223881 0.83208955 0.81716418 0.82835821 0.83208955 0.8358209 0.83208955] with the max value as 0.835820895522388
Gaussian Naive Bayes
model=GaussianNB()
model.fit(train_X,train_Y)
prediction6=model.predict(test_X)
print('The accuracy of the NaiveBayes is',metrics.accuracy_score(prediction6,test_Y))>> The accuracy of the NaiveBayes is 0.8134328358208955
Random Forest
model=RandomForestClassifier(n_estimators=100)
model.fit(train_X,train_Y)
prediction7=model.predict(test_X)
print('The accuracy of the Random Forests is',metrics.accuracy_score(prediction7,test_Y))>> The accuracy of the Random Forests is 0.8134328358208955
모델의 정확도만일 분류기의 성능을 결정하는 요인은 아니다. 분류기가 train data로 훈련이 되고 test data에 대해 테스트 되었으며 정확도가 90%라고 가정해보자.
이것은 분류기 치고는 매우 좋은 정확도로 보이지만, 새로 도입되는 모든 테스트 세트에 대해 정확도가 90%가 될 것이라고 확신할 수는 없다. train 및 test 데이터가 바뀜에 다라 정확도도 바귄다. 정확도가 상승하거나 하락할수도 있다. 이를 모델의 분산이라고 한다.
이를 극복하고 일반화된 모델을 얻기 위해서는 교차 검증을 할 수 있다.
Cross Validation
교차검증은 train set을 train set + validation set으로 분리한 뒤, validation set을 사용해 검증하는 방식이다.
마치 축구 대표팀이 팀 훈련(train)을 하고 바로 월드컵(test)로 나가는 것이 아니라, 팀 훈련(train)을 한 다음 평가전(valid)를 거쳐 팀의 훈련 강도(학습 정도)를 확인하고 월드컵(test)에 나가는 것과 비슷하다.
Scikit-learn - Cross validation, K-fold 사용하기
kfold = KFold(n_splits=10, random_state=22)
xyz=[]
accuracy=[]
std=[]
classifiers=['Linear Svm','Radial Svm','Logistic Regression','KNN','Decision Tree','Naive Bayes','Random Forest']
models=[svm.SVC(kernel='linear'),svm.SVC(kernel='rbf'),LogisticRegression(),KNeighborsClassifier(n_neighbors=9),DecisionTreeClassifier(),GaussianNB(),RandomForestClassifier(n_estimators=100)]
for i in models:
model = i
cv_result = cross_val_score(model,X,Y, cv = kfold,scoring = "accuracy")
cv_result=cv_result
xyz.append(cv_result.mean())
std.append(cv_result.std())
accuracy.append(cv_result)
new_models_dataframe2=pd.DataFrame({'CV Mean':xyz,'Std':std},index=classifiers)
new_models_dataframe2
plt.subplots(figsize=(12,6))
box=pd.DataFrame(accuracy,index=[classifiers])
box.T.boxplot()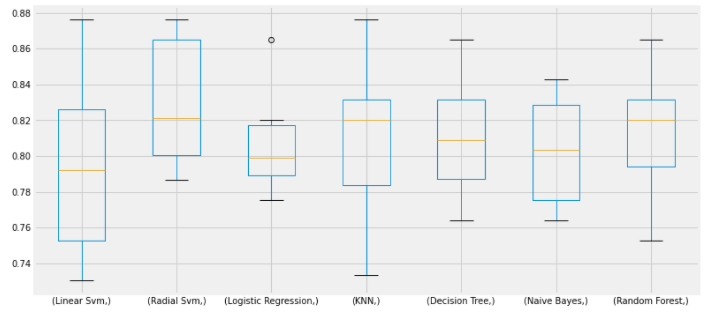
new_models_dataframe2['CV Mean'].plot.barh(width=0.8)
plt.title('Average CV Mean Accuracy')
fig=plt.gcf()
fig.set_size_inches(8,5)
plt.show()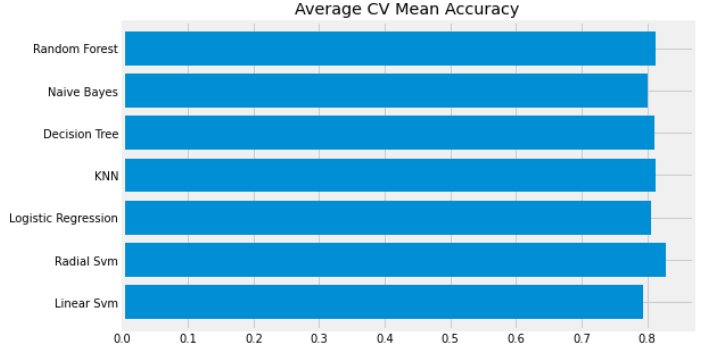
Confusion Matrix
때로는 자료의 불균형으로 인해 분류의 정확도가 잘못될 수도 있다. 우리는 혼동행렬로 요약된 결과를 얻을 수 있는데, 이것은 모델이 어디에서 잘못되었는지 또는 모델이 어떤 클래스를 잘못예측했는지 보여준다.
y_pred = cross_val_predict(svm.SVC(kernel='rbf'),X,Y,cv=10)
sns.heatmap(confusion_matrix(Y,y_pred),ax=ax[0,0],annot=True,fmt='2.0f')
Hyper-Parameters Tuning
SVM
from sklearn.model_selection import GridSearchCV
C=[0.05,0.1,0.2,0.3,0.25,0.4,0.5,0.6,0.7,0.8,0.9,1]
gamma=[0.1,0.2,0.3,0.4,0.5,0.6,0.7,0.8,0.9,1.0]
kernel=['rbf','linear']
hyper={'kernel':kernel,'C':C,'gamma':gamma}
gd=GridSearchCV(estimator=svm.SVC(),param_grid=hyper,verbose=True)
gd.fit(X,Y)
print(gd.best_score_)
print(gd.best_estimator_)>> 0.8282593685267716
SVC(C=0.4, break_ties=False, cache_size=200, class_weight=None, coef0=0.0, decision_function_shape='ovr', degree=3, gamma=0.3, kernel='rbf', max_iter=-1, probability=False, random_state=None, shrinking=True, tol=0.001, verbose=False)
Random Foreset
n_estimators=range(100,1000,100)
hyper={'n_estimators':n_estimators}
gd=GridSearchCV(estimator=RandomForestClassifier(random_state=0),param_grid=hyper,verbose=True)
gd.fit(X,Y)
print(gd.best_score_)
print(gd.best_estimator_)>> 0.819327098110602
RandomForestClassifier(bootstrap=True, ccp_alpha=0.0, class_weight=None, criterion='gini', max_depth=None, max_features='auto', max_leaf_nodes=None, max_samples=None, min_impurity_decrease=0.0, min_impurity_split=None, min_samples_leaf=1, min_samples_split=2, min_weight_fraction_leaf=0.0, n_estimators=300, n_jobs=None, oob_score=False, random_state=0, verbose=0, warm_start=False)
rbf-SVM의 최고 점수는 C=0.05, gamma=0.1 일때 82.82%이다. randomforest는 n_estimators=900일때 약 81.8%이다.
Ensembling
앙상블은 모델의 정확도나 성능을 높이는 좋은 방법이다. 간단히 말해 하나의 강력한 모델을 만들기 위해 다양한 간단한 모델을 조합하는 것이다.
Voting Classifier
from sklearn.ensemble import VotingClassifier
ensemble_lin_rbf=VotingClassifier(estimators=[('KNN',KNeighborsClassifier(n_neighbors=10)),
('RBF',svm.SVC(probability=True,kernel='rbf',C=0.5,gamma=0.1)),
('RFor',RandomForestClassifier(n_estimators=500,random_state=0)),
('LR',LogisticRegression(C=0.05)),
('DT',DecisionTreeClassifier(random_state=0)),
('NB',GaussianNB()),
('svm',svm.SVC(kernel='linear',probability=True))
],
voting='soft').fit(train_X,train_Y)
print('The accuracy for ensembled model is:',ensemble_lin_rbf.score(test_X,test_Y))
cross=cross_val_score(ensemble_lin_rbf,X,Y, cv = 10,scoring = "accuracy")
print('The cross validated score is',cross.mean())>> The accuracy for ensembled model is: 0.8208955223880597
>> The cross validated score is 0.8249188514357053
Bagging
Bagged KNN
from sklearn.ensemble import BaggingClassifier
model=BaggingClassifier(base_estimator=KNeighborsClassifier(n_neighbors=3),random_state=0,n_estimators=700)
model.fit(train_X,train_Y)
prediction=model.predict(test_X)
print('The accuracy for bagged KNN is:',metrics.accuracy_score(prediction,test_Y))
result=cross_val_score(model,X,Y,cv=10,scoring='accuracy')
print('The cross validated score for bagged KNN is:',result.mean())>> The accuracy for bagged KNN is: 0.835820895522388
>> The cross validated score for bagged KNN is: 0.8160424469413232
Bagged Decision Tree
model=BaggingClassifier(base_estimator=DecisionTreeClassifier(),random_state=0,n_estimators=100)
model.fit(train_X,train_Y)
prediction=model.predict(test_X)
print('The accuracy for bagged Decision Tree is:',metrics.accuracy_score(prediction,test_Y))
result=cross_val_score(model,X,Y,cv=10,scoring='accuracy')
print('The cross validated score for bagged Decision Tree is:',result.mean())>> The accuracy for bagged Decision Tree is: 0.8246268656716418
>> The cross validated score for bagged Decision Tree is: 0.8227590511860174
Boosting
AdaBoost(Adaptive Boosting)
from sklearn.ensemble import AdaBoostClassifier
ada=AdaBoostClassifier(n_estimators=200,random_state=0,learning_rate=0.1)
result=cross_val_score(ada,X,Y,cv=10,scoring='accuracy')
print('The cross validated score for AdaBoost is:',result.mean())>> The cross validated score for AdaBoost is: 0.8249188514357055
Stochastic Gradient Boosting
from sklearn.ensemble import GradientBoostingClassifier
grad=GradientBoostingClassifier(n_estimators=500,random_state=0,learning_rate=0.1)
result=cross_val_score(grad,X,Y,cv=10,scoring='accuracy')
print('The cross validated score for Gradient Boosting is:',result.mean())>> The cross validated score for Gradient Boosting is: 0.8115230961298376
XGBoost
import xgboost as xg
xgboost=xg.XGBClassifier(n_estimators=900,learning_rate=0.1)
result=cross_val_score(xgboost,X,Y,cv=10,scoring='accuracy')
print('The cross validated score for XGBoost is:',result.mean())>> The cross validated score for XGBoost is: 0.8115480649188515
Hyper-Parameter Tuning for AdaBoost
n_estimators=list(range(100,1100,100))
learn_rate=[0.05,0.1,0.2,0.3,0.25,0.4,0.5,0.6,0.7,0.8,0.9,1]
hyper={'n_estimators':n_estimators,'learning_rate':learn_rate}
gd=GridSearchCV(estimator=AdaBoostClassifier(),param_grid=hyper,verbose=True)
gd.fit(X,Y)
print(gd.best_score_)
print(gd.best_estimator_)>> 0.8293892411022534
>> AdaBoostClassifier(algorithm='SAMME.R', base_estimator=None, learning_rate=0.1, n_estimators=100, random_state=None)
AdaBoost로 얻을 수 있는 최대 정확도는 n_contactator=200, learning_rate=0.05 일 때 83.16%이다.
Feature Importance
학습된 모델은 feature importance를 가지게 되는데, 이것을 확인해서 지금 만든 모델이 어떤 feature에 영향을 많이 받았는지 확인할 수 있다.
학습된 모델은 기본적으로 feature importance를 가지고 있어서 쉽게 그 수치를 얻을 수 있다.
f,ax=plt.subplots(2,2,figsize=(15,12))
model=RandomForestClassifier(n_estimators=500,random_state=0)
model.fit(X,Y)
pd.Series(model.feature_importances_,X.columns).sort_values(ascending=True).plot.barh(width=0.8,ax=ax[0,0])
ax[0,0].set_title('Feature Importance in Random Forests')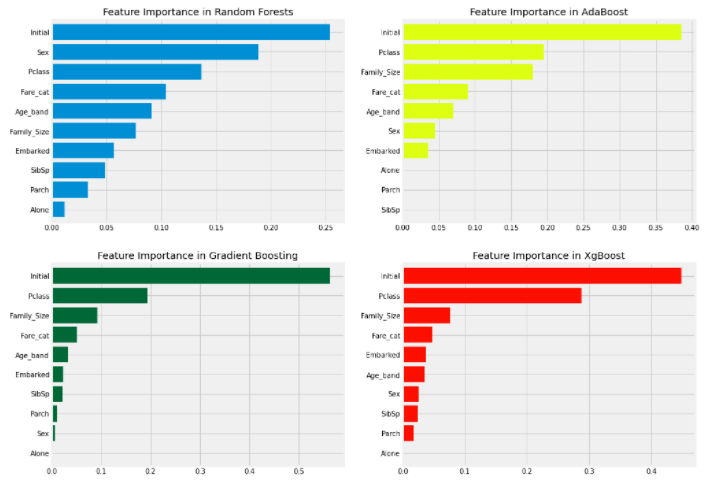

끝입니다!!!
Reference
https://kongdols-room.tistory.com/172
https://workingwithpython.com/howtohandlemissingvaluewithpython/
https://m.blog.naver.com/PostView.naver?isHttpsRedirect=true&blogId=youji4ever&logNo=221623491491
https://kongdols-room.tistory.com/174
https://junklee.tistory.com/10
https://ponyozzang.tistory.com/291
https://3months.tistory.com/194
https://m.blog.naver.com/youji4ever/221843477880
https://m.blog.naver.com/PostView.naver?isHttpsRedirect=true&blogId=youji4ever&logNo=221804922659
https://blog.naver.com/PostView.nhn?blogId=youji4ever&logNo=222292901767&parentCategoryNo=&categoryNo=&viewDate=&isShowPopularPosts=false&from=postView
https://dojang.io/mod/page/view.php?id=2360
https://blog.naver.com/PostView.nhn?blogId=youji4ever&logNo=221864110030
https://m.blog.naver.com/youji4ever/221791455668
https://bearwoong.tistory.com/65
https://zephyrus1111.tistory.com/91
https://hongl.tistory.com/89
https://notebook.community/zzsza/Datascience_School/13. Scikit-Learn%2C Statsmodel/01. Scikit-Learn 패키지의 소개
'심화 스터디 > 코드 분석 스터디' 카테고리의 다른 글
| [코드 분석 스터디] Binary Classification: Image Classfication - Statoil/C-CORE Iceberg Classifier Challenge (2) | 2021.09.25 |
|---|---|
| [코드 분석 스터디] Multi-class classification : Image classification - TensorFlow Speech Recognition Challenge (2) | 2021.09.23 |
| [코드 분석 스터디] Porto Seguro's Safe Driver Prediction 캐글 커널 필사 (3) | 2021.09.16 |
| [코드 분석 스터디] 스터디 진행 계획 및 방법 (0) | 2021.09.09 |
| 코드 분석 스터디 소개 (0) | 2021.08.27 |



댓글 영역