상세 컨텐츠
본문 제목
[코드 분석 스터디] Binary Classification: Image Classfication - Statoil/C-CORE Iceberg Classifier Challenge
본문
참고 커널:
https://www.kaggle.com/sh0wmaker/binary-image-classification
0. 대회 소개
대회에 대한 간략한 소개:
북해에서 항해를 하다보면 맞닥드리는 제일 위험한 상황은 표류하는 빙하와의 충돌이다. 이 문제에 대해 골머리를 앓고 있던 에너지 회사인 Statoil사는 위성 회사인 C-CORE와 협력을 하여 표류 빙하와 일반적인 배의 위성 사진을 분류하는 모델을 만들기 위해 이 대회를 개최하게 되었다.
데이터 소개
데이터는 지상 600km에 위치한 위성에서 레이더를 쏴 반사된 빛을 이용해 이미지화(backscatter)한 것이라 한다. 물체가 견고할수록(land, islands, sea ice, icebergs, ships) 더 강한 레이더 에너지가 반사되어 실제 이미지에서 더 밝게 나온다고 하며, 이 원리로 바다에 떠있는 견고한 물체인 배나 빙하를 식별할 수 있다고 한다. 레이더는 수직, 수평 두 방향으로 쏘는데, 각 방향으로 생성된 이미지는 band_1, band_2에 저장되어있다.

데이터의 Features:
1. id : 이미지 id
2. band_1, band_2 : 레이더 1, 2에서 backscatter로 생성된 이미지 데이터이며 2차원 데이터를 1차원으로 flatten한 데이터
3. inc_angle : 이미지가 찍한 각도
4. is_iceberg : 1 = 빙하, 0 = 배 를 나타내는 Target 변수
채점 방식
Log Loss (cross entropy) 사용
1. Data Exploration
train.head(5)
- DataFrame.head() : DataFrame내의 처음 5줄의 데이터를 출력하며, 각 열의 값에 대해 간단하게 살펴볼때 유용한 메서드
이미지 데이터인 band_1, band_2를 보면 1차원 리스트로 되어있는것을 확인할 수 있으며, 나중에 2차원 데이터로 변경해줘야 한다.
train['inc_angle'].value_counts()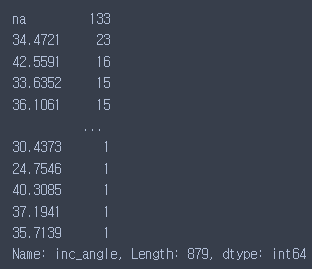
- list.value_counts() : 리스트에서 등장하는 모든 값과 그 값들의 빈도수를 보여주는 메서드
출력 결과를 살펴보면 결측값를 뜻하는 'na'가 133개나 존재하는 것을 알 수 있는데, 이 문제를 해결하기위해 이 커널에서는 결측치를 모두 0으로 바꿔주는 방법을 사용했다.
train.inc_angle = train.inc_angle.replace('na',0)
train.inc_angle = train.inc_angle.astype(float).fillna(0.0)- list.replace(a, b) : list에 등장하는 모든 a값을 b값으로 변경해주는 메서드
- list.astype(type) : 리스트의 모든 아이템을 type 타입으로 바꿔주는 메서드
'na'값을 제외한 다른 값들이 모두 float형이기 때문에 우리가 넣어준 0 역시 float형으로 변경해준다
len(train.loc[0,'band_1'])- len(list) : list의 길이를 반환해주는 메서드
앞서 설명했다시피 band_1과 band_2는 이미지 데이터이기 떄문에 75 x 75 크기의 2차원 데이터로 변환을 해준다.
X_band_1 = np.array([np.array(band).astype(np.float32).reshape(75, 75) for band in train['band_1']]);
X_band_2 = np.array([np.array(band).astype(np.float32).reshape(75, 75) for band in train['band_2']]);- np.array.reshape() : numpy array의 차원을 바꿔주는 메서드. 변경전과 후의 총 입력 cell의 수는 달라지면 안됨
우리는 지금 두개의 2차원 이미지 데이터를 가지고 있지만, 하나의 딥러닝 모델로 학습를 시켜려면 그 두개의 데이터를 하나의 데이터로 합쳐줘야한다. 그래서 컬러사진의 RGB값이 세개의 채널을 가지고 있는 것 처럼 X_band_1을 채널1, X_band_2를 채널2 마지막으로 두개의 채널을 더해서 2로 나눠준 값을 채널3으로 설정해서 마치 하나의 컬러 이미지를 생성하는 것과 같은 방식으로 두개의 채널을 하나의 이미지 데이터로 이어주었다.
X_train = np.concatenate([X_band_1[:, :, :, np.newaxis],
X_band_2[:, :, :, np.newaxis],
((X_band_1+X_band_2)/2)[:, :, :, np.newaxis]],
axis=-1)- np.array.concatenate([list1, list2, ..., listn], axis) : axis값에 따라 특정한 축으로 여러개의 리스트를 이어붙혀주는 메서드
Test 데이터에도 동일하게 적용.
X_band_test_1=np.array([np.array(band).astype(np.float32).reshape(75, 75) for band in test["band_1"]])
X_band_test_2=np.array([np.array(band).astype(np.float32).reshape(75, 75) for band in test["band_2"]])
X_test = np.concatenate([X_band_test_1[:, :, :, np.newaxis]
, X_band_test_2[:, :, :, np.newaxis]
, ((X_band_test_1+X_band_test_2)/2)[:, :, :, np.newaxis]], axis=-1)2. Data Visualization
train.loc[14, 'is_iceberg']
# 출력 : 0우리가 시각화에 사용할 14번 데이터는 ship 데이터이다. Plotly를 사용해서 3D 시각화를 한번 해보면:
c, name = X_band_1[14, :, :], 'Ship' # c에 14번 데이터 저장
data = [go.Surface(z = c)] # z축값을 c의값으로 설정
layout = go.Layout() # 레이아웃 세팅
fig = go.Figure(data=data, layout=layout)
py.iplot(fig)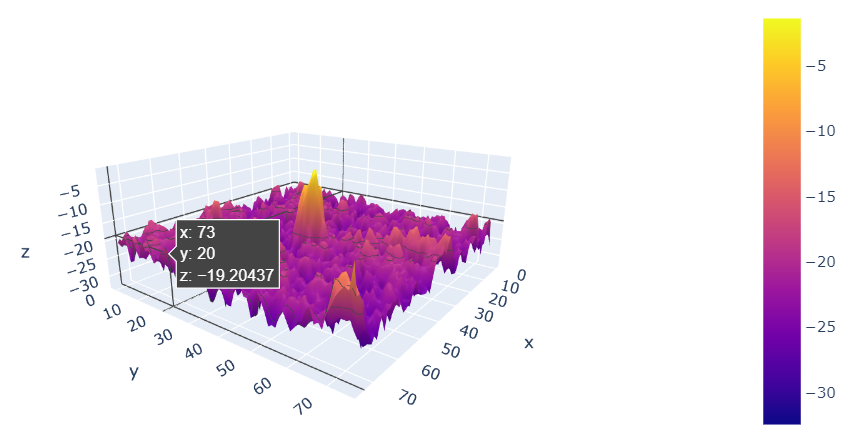
위와 같은 Plotly 시각화 툴이 나타나게 되는데, Plotly 시각화의 특징은 사용자가 마우스를 활용해 보는 각도를 조절하거나 특정한 위치의 x, y, z 값들을 보는 등 interactive한 시각화를 해준다는 것이다.
다음은 서로 다른 각도(band_1, band_2)에서 같은 물체를 찍었을 때 차이점을 시각화하는 코드이다:
label = 'ship'
#같은 14번 ship이지만 보는 각에 따라서 나오는 것이 다르다는 것을 보여주는 것
band1 = X_band_1[14,:,:]
band2 = X_band_2[14,:,:]
#subplot메서드처럼 여러개의 시각화를 하나의 출력으로 보여주고 싶다면 Plotly.tools를 사용해야 함
fig = tools.make_subplots(rows=1,cols=2, specs=[[{'is_3d': True}, {'is_3d':True}]]) # 1행, 2열의 팔레트 생성
data = go.Surface(z = band1, colorscale='RdBu_r', scene='scene1', showscale=True)
data1 = go.Surface(z = band2, colorscale='RdBu_r', scene='scene1', showscale=True)
fig['layout'].update(title='3D surface plot for "{}" (left is from band1, right is from band2)'.format(label), titlefont=dict(size=20), height=600, width=800)
fig.append_trace(data,1,1)
fig.append_trace(data1,1,2)
py.iplot(fig)
두 자료 모두 14번 데이터를 시각화한 것이지만, 왼쪽은 band_1 데이터, 오른쪽은 band_2 데이터인데 시각화를 해봤을때 확실히 이미지가 찍힌 각도에 따라 같은 물체도 다르게 보이는 것을 볼 수 있다.
이미지 데이터는 z축 방면에서 bird's eye view로 보게되면 2차원 이미지 데이터로도 볼 수 있는데, matplotlib의 imshow() 함수를 사용하면 간단하게 가능하다:
plt.imshow(band1)
plt.show()
3. Modeling
(처음 보시는 분들을 위한) 딥러닝이란?
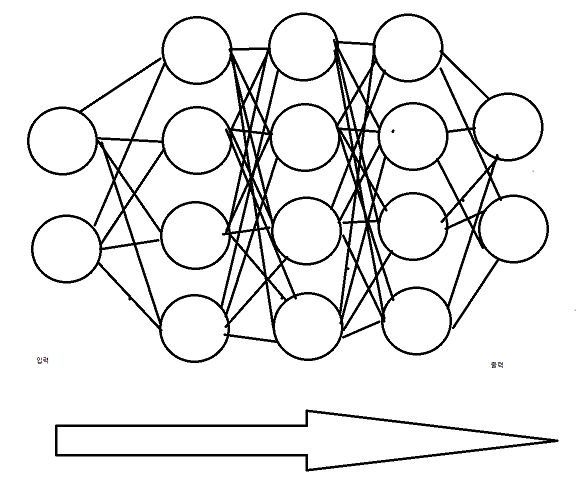
학습을 진행할 때 몇개의 층을 만드는데, 각 층마다 뉴런들이 존재한다. 그리고 각 뉴런들은 자신에게 들어온 신호를 가중치와 곱해 모두 더하고, 역치와 비교해서 신호를 다음 층의 뉴런으로 전달하는 방법이다. 3개 이상의 레이어를 쌓으면 딥러닝이라고 한다.
CNN에 대한 간단한 설명
CNN은 인간의 시신경 구조를 모방해 만들어진 인공신경망(딥러닝) 알고리즘이다. 다수의 Convolutional Layer(필터)로 부터 특징맵(Feature map)을 추출하고 서브샘플링(Subsampling)을 통해 차원을 축소하여 특징맵에서 중요한 부분만을 가져온다.
사용할 레이어:
Conv2D:

- Convolution Filiter을 이용하여 이미지의 특징을 추출 (이때 하나만 만드는 것이 아니라 여러 개의 filter을 이용하여 여러 특징을 추출)
- filter는 학습된 weight 값으로 보통 크기(ex. 3 X 3)만 지정함
- 한칸씩 위치를 옮겨가며 특징을 추출한다고 함
MaxPooling:
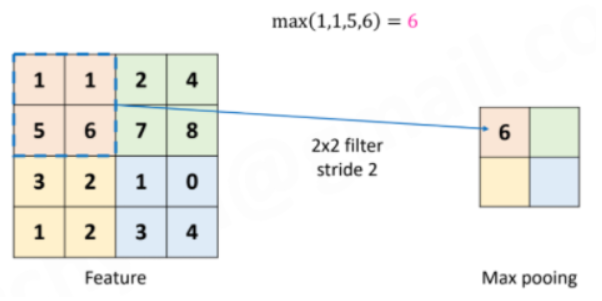
사용자가 정의한 크기만큼의 영역에서 최대의 특징값을 기록한다. MaxPooling Layer를 사용하는 이유는:
- input size를 줄임. : 여러번 convolution layer을 반복하게 되는데, 별로 필요하지 않은 자료까지 전부를 다 분석할 필요가 없다.
- overfitting을 조절 : input size가 줄어드는 것은 그만큼 쓸데없는 parameter의 수가 줄어들며 Overfitting을 방지할 수 있다.
그 외 사용한 레이어:
Dense : 딥러닝의 가장 기본적인 레이어로써 입력과 출력을 모두 연결해주며, 입출력을 각각 연결해주는 가중치를 포함하고 있다
Activation : Dense 레이어에서는 보통 신호에 가중치를 곱하는 선형적인 방식으로 연결하지만, Activation 함수를 사용하면 sigmoid, tanh등 다른 방법으로 연결 가능하다.
Dropout : Overfitting을 방지하기 위한 레이어이며 hidden layer의 일부 유닛이 동작하지 않게 하는 방법
Flatten : 다차원의 입력 tensor을 1-차원으로 변경해주는 레이어
우리가 만들 모델의 대략적인 구조

#define our model
def getModel():
#Inifializing a model
gmodel=Sequential()
#Conv Layer 1
gmodel.add(Conv2D(64, kernel_size=(3, 3),activation='relu', input_shape=(75, 75, 3)))
gmodel.add(MaxPooling2D(pool_size=(3, 3), strides=(2, 2)))
gmodel.add(Dropout(0.2))
#Conv Layer 2
gmodel.add(Conv2D(128, kernel_size=(3, 3), activation='relu' ))
gmodel.add(MaxPooling2D(pool_size=(2, 2), strides=(2, 2)))
gmodel.add(Dropout(0.2))
#Conv Layer 3
gmodel.add(Conv2D(128, kernel_size=(3, 3), activation='relu'))
gmodel.add(MaxPooling2D(pool_size=(2, 2), strides=(2, 2)))
gmodel.add(Dropout(0.2))
#Conv Layer 4
gmodel.add(Conv2D(64, kernel_size=(3, 3), activation='relu'))
gmodel.add(MaxPooling2D(pool_size=(2, 2), strides=(2, 2)))
gmodel.add(Dropout(0.2))
#Flatten the data for upcoming dense layers
gmodel.add(Flatten())
#Dense Layers
gmodel.add(Dense(512))
gmodel.add(Activation('relu'))
gmodel.add(Dropout(0.2))
#Dense Layer 2
gmodel.add(Dense(256))
gmodel.add(Activation('relu'))
gmodel.add(Dropout(0.2))
#Sigmoid Layer
gmodel.add(Dense(1))
gmodel.add(Activation('sigmoid'))
mypotim=Adam(lr=0.001, beta_1=0.9, beta_2=0.999, epsilon=1e-08, decay=0.0)
gmodel.compile(loss='binary_crossentropy',
optimizer=mypotim,
metrics=['accuracy'])
gmodel.summary()
return gmodel
def get_callbacks(filepath, patience=2):
es = EarlyStopping('val_loss', patience=patience, mode="min")
msave = ModelCheckpoint(filepath, save_best_only=True) # best model configuration을 msave에 저장해준다
return [es, msave]
file_path = ".model_weights.hdf5"
callbacks = get_callbacks(filepath=file_path, patience=5)Keras에서는 딥러닝 모델 building 방법이 두 가지가 있는데 Sequential 방식과 Functional 방식이다. Sequential 형태의 model building은 먼저 생성해둔 모델에 model.add(layer)와 같은 방식으로 layer 객체를 add 메서드의 파라미터로 넘겨주면 모델에 해당 레이어가 마치 블록을 쌓듯이 쌓이는 구조이다. Functional model은 add 메서드를 쓰는 대신, 레이어를 생성하면 함수가 반환되는데 그 함수에 모델을 argument로 넣어주어 새로운 모델 변수에 반환된 함수의 반환값을 저장하는 방식이다. 여기서 반환값은 원래 모델에 레이어가 합쳐진 모델이다. 김종민님의 muli-class classification 포스팅에 Functional 모델을 사용하니 한번 참고하면 좋을 것 같다. 그렇게 model building이 완료되면 딥러닝 모델은 꼭 compile을 해줘야 한다고 한다!
get_callbacks함수에서 EarlyStopping(monitor, patience, mode) 함수는 이름과 같이 Early Stopping을 통해 모델이 Overfitting되는 것을 방지하는데, monitor 인자는 EarlyStopping의 기준이 되는, mode는 min인지 max인지 정하는, 마지막으로 patience는 몇 epoch 동안 minimize가 안되면 학습을 멈출것인지 기준을 정하는 인자라고 한다. 그렇게 EarlyStopping가 실행이 되면 최선의 model configuration을 msave 변수에 저장을 해주고, 최종적으로 그 값을 반환해주는 것이 get_callbacks함수의 목적이다.
모델 생성 함수를 생성했으니 이제 모델 훈련과 평가만 남았다.
target_train = train['is_iceberg']
X_train, X_valid, y_train, y_valid = train_test_split(X_train, target_train, random_state=1, train_size = 0.8)train_test_split으로 train set과 validation set으로 나눠준 후
SEED = 2021
np.random.seed(SEED)
tf.random.set_seed(SEED)일관성있게 모델 성능을 평가하기 위해 랜덤 시드 값을 고정했다. 이제 드디어 모델을 불러오고 훈련을 실시할 차례이다.
gmodel=getModel() # gmodel에 미리 만들어두었던 모델을 생성 및 저장
gmodel.fit(X_train, y_train,
batch_size=24,
epochs=10,
verbose=1,
validation_data=(X_valid, y_valid),
callbacks=callbacks) # 모델 학습 시작Model: "sequential_1"
_________________________________________________________________
Layer (type) Output Shape Param #
=================================================================
conv2d (Conv2D) (None, 73, 73, 64) 1792
_________________________________________________________________
max_pooling2d (MaxPooling2D) (None, 36, 36, 64) 0
_________________________________________________________________
dropout (Dropout) (None, 36, 36, 64) 0
_________________________________________________________________
conv2d_1 (Conv2D) (None, 34, 34, 128) 73856
_________________________________________________________________
max_pooling2d_1 (MaxPooling2 (None, 17, 17, 128) 0
_________________________________________________________________
dropout_1 (Dropout) (None, 17, 17, 128) 0
_________________________________________________________________
conv2d_2 (Conv2D) (None, 15, 15, 128) 147584
_________________________________________________________________
max_pooling2d_2 (MaxPooling2 (None, 7, 7, 128) 0
_________________________________________________________________
dropout_2 (Dropout) (None, 7, 7, 128) 0
_________________________________________________________________
conv2d_3 (Conv2D) (None, 5, 5, 64) 73792
_________________________________________________________________
max_pooling2d_3 (MaxPooling2 (None, 2, 2, 64) 0
_________________________________________________________________
dropout_3 (Dropout) (None, 2, 2, 64) 0
_________________________________________________________________
flatten (Flatten) (None, 256) 0
_________________________________________________________________
dense (Dense) (None, 512) 131584
_________________________________________________________________
activation (Activation) (None, 512) 0
_________________________________________________________________
dropout_4 (Dropout) (None, 512) 0
_________________________________________________________________
dense_1 (Dense) (None, 256) 131328
_________________________________________________________________
activation_1 (Activation) (None, 256) 0
_________________________________________________________________
dropout_5 (Dropout) (None, 256) 0
_________________________________________________________________
dense_2 (Dense) (None, 1) 257
_________________________________________________________________
activation_2 (Activation) (None, 1) 0
=================================================================
Total params: 560,193
Trainable params: 560,193
Non-trainable params: 0
_________________________________________________________________
Epoch 1/10
54/54 [==============================] - 9s 155ms/step - loss: 0.9423 - accuracy: 0.5269 - val_loss: 0.5923 - val_accuracy: 0.6449
Epoch 2/10
54/54 [==============================] - 8s 151ms/step - loss: 0.5744 - accuracy: 0.6539 - val_loss: 0.5644 - val_accuracy: 0.7072
Epoch 3/10
54/54 [==============================] - 8s 150ms/step - loss: 0.5003 - accuracy: 0.7405 - val_loss: 0.4758 - val_accuracy: 0.7944
Epoch 4/10
54/54 [==============================] - 8s 151ms/step - loss: 0.4637 - accuracy: 0.7732 - val_loss: 0.4752 - val_accuracy: 0.7477
Epoch 5/10
54/54 [==============================] - 8s 153ms/step - loss: 0.4748 - accuracy: 0.7677 - val_loss: 0.4447 - val_accuracy: 0.8193
Epoch 6/10
54/54 [==============================] - 8s 151ms/step - loss: 0.4135 - accuracy: 0.8106 - val_loss: 0.3815 - val_accuracy: 0.8349
Epoch 7/10
54/54 [==============================] - 8s 150ms/step - loss: 0.4220 - accuracy: 0.8098 - val_loss: 0.4008 - val_accuracy: 0.8224
Epoch 8/10
54/54 [==============================] - 8s 151ms/step - loss: 0.4193 - accuracy: 0.8020 - val_loss: 0.3699 - val_accuracy: 0.8349
Epoch 9/10
54/54 [==============================] - 8s 150ms/step - loss: 0.4103 - accuracy: 0.7942 - val_loss: 0.4469 - val_accuracy: 0.8162
Epoch 10/10
54/54 [==============================] - 8s 150ms/step - loss: 0.4228 - accuracy: 0.7942 - val_loss: 0.3634 - val_accuracy: 0.8380
<keras.callbacks.History at 0x1fab07e97f0>model.history.history애 접근하면 epoch를 거듭하며 저장한 loss 값을 확인할 수 있다.
hist = gmodel.history
plt.plot(hist.history['val_loss'])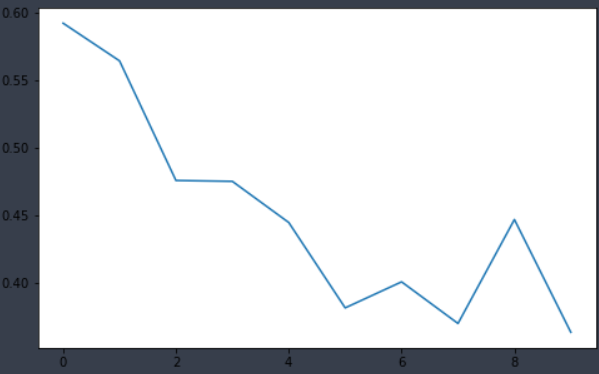
gmodel.load_weights(filepath=file_path)
score = gmodel.evaluate(X_valid, y_valid, verbose=1)
print('Test loss:', score[0])
print('Test accuracy:', score[1])
# 출력 #
#11/11 [==============================] - 0s 40ms/step - loss: 0.3634 - accuracy: 0.8380
#Test loss: 0.3633979558944702
#Test accuracy: 0.8380062580108643



댓글 영역